Quand vous inférez un arbre phylogénétique à partir d'un gène (ou d'une protéine), vous produisez l'arbre phylogénétique de ce gène qui, si cet arbre est robuste et fiable, raconte l'histoire évolutive de ce gène. Dans les cas d'histoire évolutives les plus simples, l'arbre phylogénétique de ce gène sera congruent avec l'arbre phylogénétique des espcès (l'abre de référence = l'arbre de la vie).
Si vous observez un arbre phylogénétique de votre gène "tout mélangé", c'est à dire où les origines taxonomiques de ce gène ne se regroupent pas en des branches homogènes, il est possible que vous soyez en présence d'un gène avec une histoire évolutive comprenant au moins un évènement de duplication. Dans le diagramme suivant, l'arbre des espèces est à gauche (A) et l'arbre du gène est à droite (B):
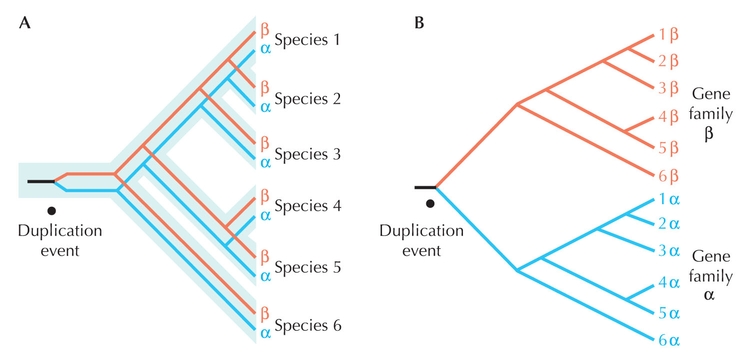
Dans l'arbre du gène à doite, les origines taxonomiques ne sont pas regroupées, car la duplication est plus ancienne que les spéciations! Ce type d'arbre où l'on voit parfaitement dans l'example ci-dessus un effet "miroir" (on voit deux arbres de phylogénie des espèces qui descendent du noeud correspondant à l'évènement de duplication) est quelque fois largement compliqué du fait que quelques fois un des deux paralogues aura été perdu dans certains clades, ou que vous aurez omis de toujours inclure les deux paralogues du gène dans chaque espèce représentée. Cet effet "miroir" devient alors cryptique et difficile à cerner...
Donc si vous observez ce genre d'arbres, ce phénomène de duplication (qui induit que vos arbres contiennent à la fois des paralogues et des orthologues) pourra éventuellement être invoqué dans votre "analyse de résultats" comme hypothèse pour expliquer ce qui peut paraître comme un arbre totalement chaotique...
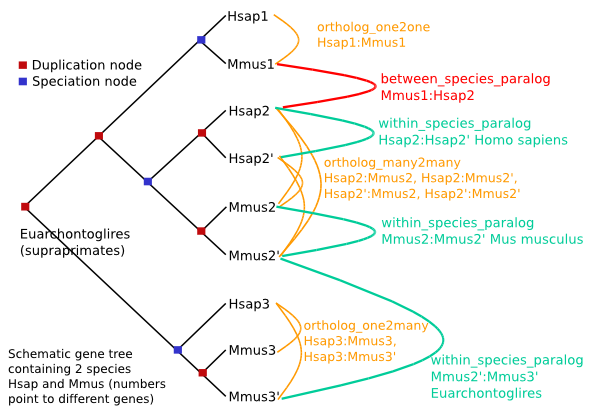
Voici un autre exemple du chaos apparent que peut induire la duplication de gènes (ici il y a seulement deux espèces, mais plusieurs duplications, notez l'apparence "mélangée" des espèces, alors que cela s'explique très bien quand on tient compte des duplications):