Annotathon: foire aux questions
Vous trouverez ici les protocoles détaillés des analyses bioinformatiques nécessaires pour l'Annotathon. Pour l'utilisation technique de l'Annotathon (création de compte, gestion du panier etc.) veuillez consulter le Manuel de l'Utilisateur. Enfin notez que ceci est un Wiki, donc tout le monde est invité à contribuer à cette documentation!
Contents
[hide]- 1 Traduction: comment repérer les ORF?
- 2 L'ORF présente-t-elle des domaines protéiques conservés?
- 3 Existe-t-il des protéines similaires (homologues) à mon ORF?
- 4 Utiliser BLAST pour compiler une liste au format FASTA des séquences homologues
- 5 L'arbre de la vie, vu par les microbes
- 6 Comment reconstruire un arbre phylogénétique?
- 7 A quoi sert le bloc Note?
Traduction: comment repérer les ORF?
- Si la région génomique ne contient aucun ORF (avec les critères arbitraires définis ici:ORF > 60 aa en particulier) => CONCLURE NON CODANT (et cocher la case située juste sous la recherche d'ORF). Dans tous les cas, avant de conclure qu'un fragment de séquence génomique est non-codant, il est conseillé d'effectuer un BLASTx de la séquence nucléotidique contre NR!
- Si la région génomique contient des ORF => ANALYSER LA PLUS
LONGUE (CRITERE 100% ARBITRAIRE). Ensuite on peut aboutir à deux cas de
figures suite à l'analyse de l'ORF la + longue :
- l'analyse de l'ORF LA PLUS LONGUE ne donne aucune similarité & aucun domaine... => Discuter si cet ORF est codant ou non (en tenant compte de la longueur de l'orf par exemple...) et cochez selon votre argumentation CODANT (par exemple si l'ORF fait quelques centaines d'AA) ou NON-CODANT. MAIS NE PAS ANALYSER LES AUTRES ORF PLUS COURTS POTENTIELLEMENT PRESENTS sur le fragment d'ADN génomique
- l'analyse de l'ORF LA PLUS LONGUE donne des résultats lors des recherches par similarité, par domaine... => conclure que l'ORF est codant (cochez CODANT et entrez les coordonnées de début et de fin de l'ORF dans l'Annotathon) et poursuivre les analyses (phylogénie etc.)
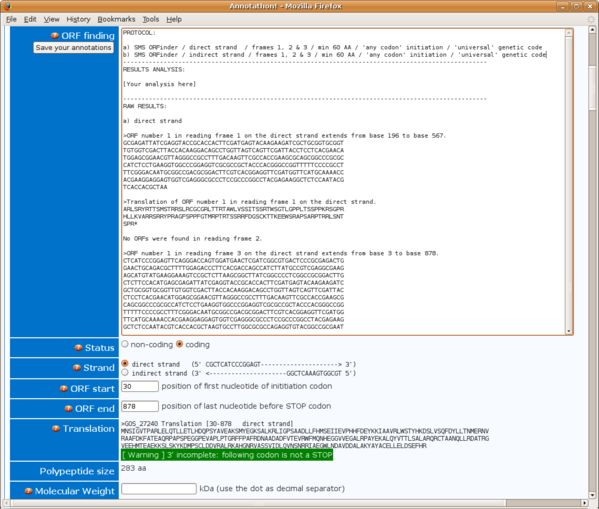
Pour ce qui est du type des codons d'initiation, commencez par une approche dite gourmande, c'est à dire celle qui produira les ORF les plus grands possibles: donc any codon
sur SMS/ORFinder. Si par la suite votre alignement multiple semble
suggérer que tous les homologues commencent plus en aval alignés sur une
Methionine interne de votre première proposition d'ORF, il est nécessaire de revoir la position de début de votre ORF pour la faire coincider avec ce qui est connu chez les protéines homologues.
Quant au choix du code génétique, réfléchissez sur le code génétique le plus probable vu l'environnement que vous analysez: est-il pertinent de choisir le code vertebrate mitochondiral...
Si vous utlisez SMS/ORFinder pour rechercher les ORF, pensez à faire l'analyse dans les cadres 1, 2 & 3 dans le sens direct puis indirect pour couvrir les 6 cadres possibles!
L'ORF présente-t-elle des domaines protéiques conservés?
Les domaines protéiques sont essentiels pour attribuer une fonction à une protéine inconnue. Si vous arrivez à indentifier un domaine protéique connu dans votre séquence, il est probable que vous seez en mesure de lui associer une fonction biochimique.
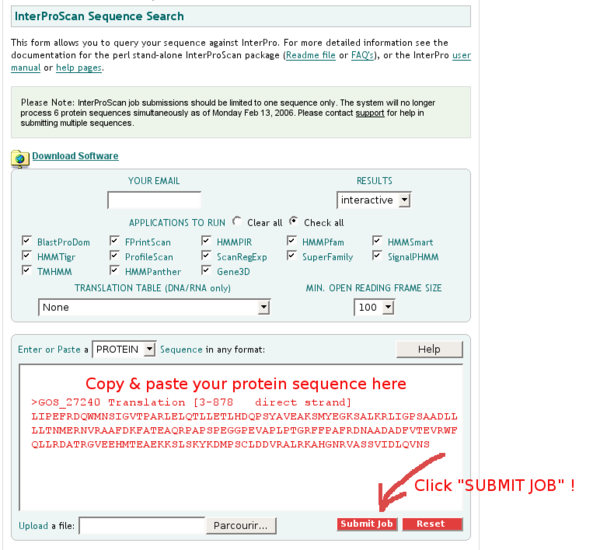
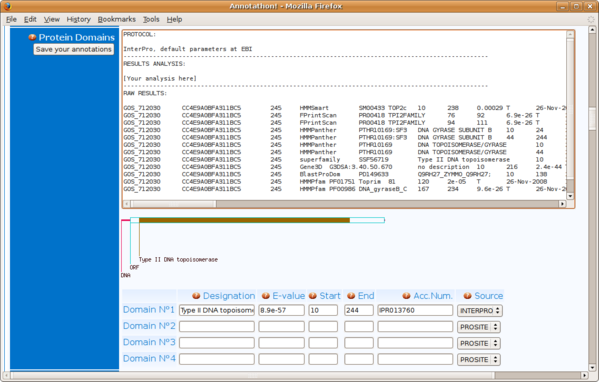
Commencez votre recherche de domaines sur le site InterproScan: il permet d'interroger la base InterPro, qui fédère un grand nombre de banques de données sur les domaines protéiques.
Après avoir cliqué sur "Submit Job", vous obtenez une page de résultats similaires à celle-ci:
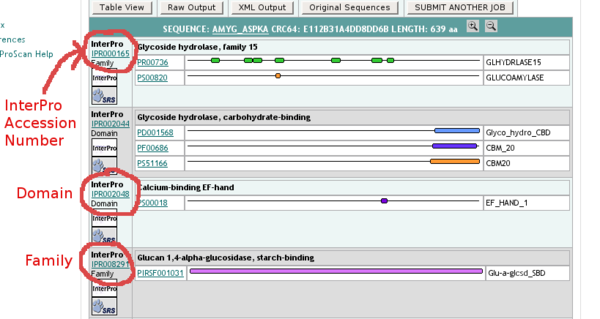
Vous obtenez une liste de domaines InterPro identifiés dans votre séquence. Attention aux points suivant:
- un domaine InterPro (p.ex. IPR000165) correspond à un ou plusieurs domaines issus d'autres banques de données (ici PR00736 issu de PRINTS et PS00820 issu de PROSITE); nous vous demandons d'indiquer le numéro d'accession InterPro (ici IPR000165)
- tous les domaines InterPro listé dans cette page ne sont pas forcément indépendants: des domaines peuvent être contenus les uns dans les autres, ou peuvent avoir des relations parent/enfant; pour trouver ces informations supplémentaires, cliquez sur "Table View" en haut de la page de résultats, pour obtenir les résultats sous cette forme
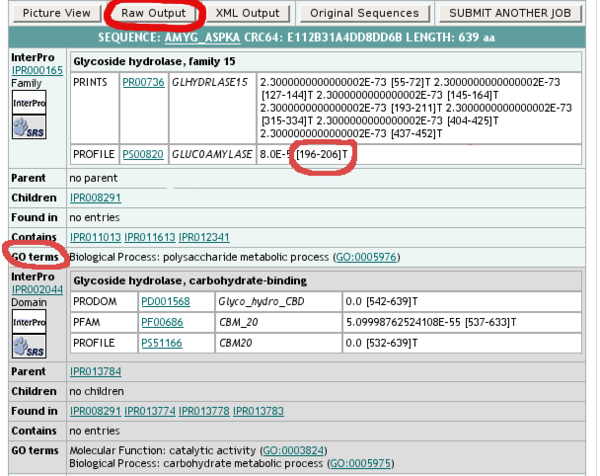
- cliquez sur le bouton "Raw output" pour avoir les résultats bruts complets au format "texte" simple, que vous copiez-collez ensuite in extenso dans le champs "Domaines: Résultats bruts" de l'Annotathon.
On voit sous la rubrique "Children" ou "contains" ou "found in" la liste des domaines InterPro reliés au domaine en question; Règle: ne listez dans le formulaire de résultat que le domaine le plus général, i.e. celui englobe les autres. Dans cet exemple, le premier domaine (IPR000165) a comme "enfant" le domaines IPR008291 (4ème dans la liste des résultats); dans ce cas, vous n'indiquez que IPR000165, et non pas IPR008291.
Vous trouvez également sur cette vue les coordonnées de début de et fin des domaines; pour le domaine IPR000165, indiquées les coordonnées extrémales du domaine PROSITE, et non pas les coordonnées de tous les petits fragments PRINTS !
Dans cette fiche apparaissent aussi des indications sur la fonction liée à ces domaines; utilisez ces informations pour attribuer une focntions biologiques (biological process ou molecular function).
Existe-t-il des protéines similaires (homologues) à mon ORF?
Recherchez d'éventuels protéines similaires à votre ORF (protéines homologues) en effectuant des BLASTs sur le site du NCBI (ou de l'EBI si le NCBI est trop lent). Les types de BLAST les plus usuels sont:
- BLASTp contre SWISSPROT (trouver des homolgues ayant des fiches bien annotées)
- BLASTp contre NR (trouver toutes les séquences similaires connues, par ex pour faire une études phylgénétique)
- BLASTx contre NR (traduit directement votre séquence génomique dans les SIX phases; uniquement si vous n'avez aucun résultat avec les BLAST ci-dessus ou si vous n'avez pas trouvé d'ORF)
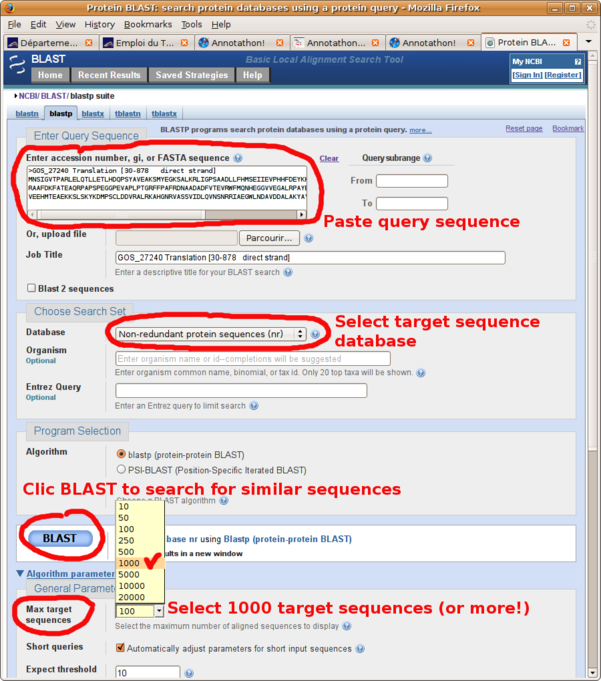
Vous commencerez toute recherche BLAST en renseignant le formulaire (Fig. B1) avec au minimum:
- la séquence requête (Query) avec un copié/collé de la séquence protéique de l'ORF pour les BLASTp et séquence ADN pour les BLASTn ou BLASTx
- la banque de données dans laquelle rechercher les homologues à la séquence requête
- sélectionnez une plus grande valeur pour Max target sequences, par exemple 500 afin d'obtenir un panorama plus large d'homologues potentiels. Si saurez qu'il faut choisir une plus grande valeur de Max target sequences quand vos derniers "hits" BLAST n'atteignent pas le limite haute fixée pour le E-value (=10 par défaut).
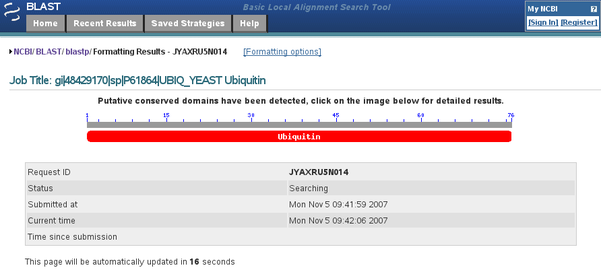
Après avoir soumis le recherche, attendez que la page d'intermède (Fig. B2) se rafraichisse et soit finalement soit remplacée par la page de résultats. Cet page intermède comporte quelque fois un diagramme coloré si des domaines protéiques conservés sont détectés dans votre séquence requête (ici un domaine 'ubiquitin'), mais il ne s'agit en aucun cas du résultat BLAST!
Entête des résultats BLAST
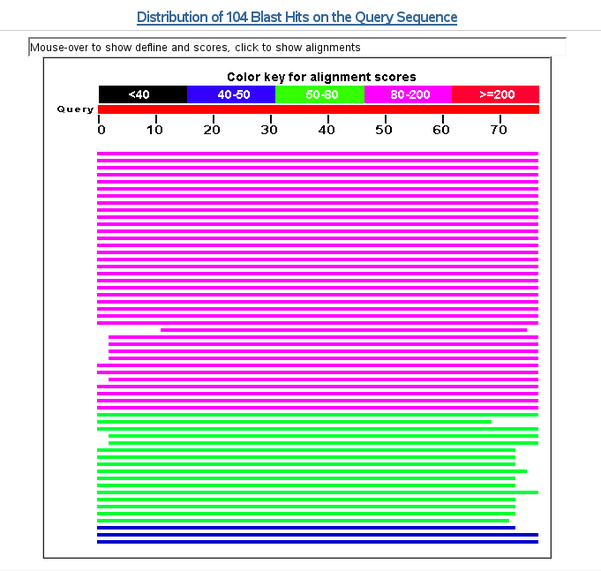
Graphique synoptique des résultats BLAST au NCBI
Liste des séquences détectées par le BLAST
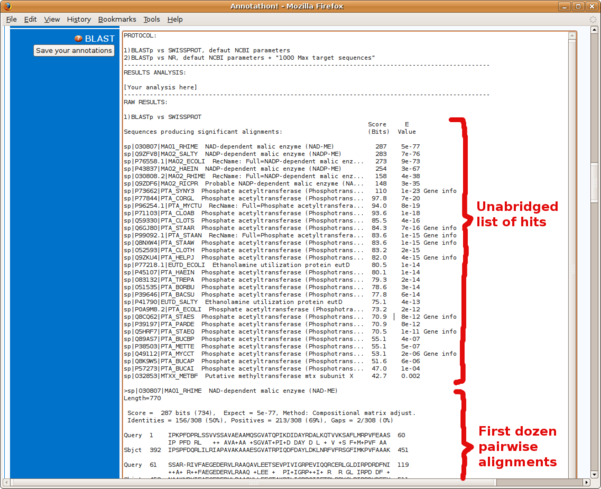
Alignements 2 à 2 des résultats BLAST
Vous reporterez obligatoirement dans l'Annotathon (Fig. B6):
- une entête résumant quelle analyse a été effectuée (ex: BLASTp de l'ORF de 231 AA contre SWISSPROT)
- la totalité du résumé des similarités détectées (Fig. B4)
- les 10 premiers alignements 2 à 2 (Fig. B5)
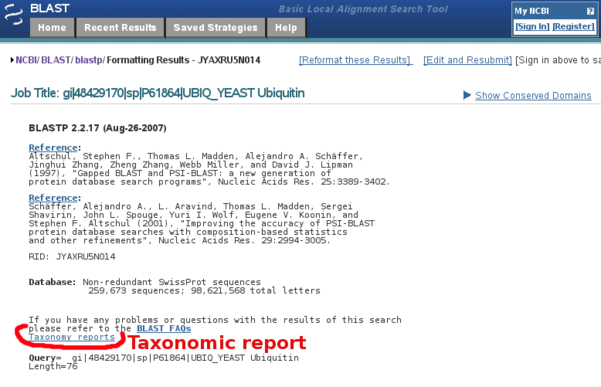
- le rapport taxonomique (la première partie Lineage Report Fig B7)
Rapport taxonomique du BLAST au NCBI (Lineage report)
Si vous faites plusieurs BLAST (par exemple un contre SP et un
contre NR), mettez-les les uns à la suite des autres dans le champ BLAST
de l'Annotathon en les séparant par exemple par une ligne de tirets
(-------------------------------------------------)
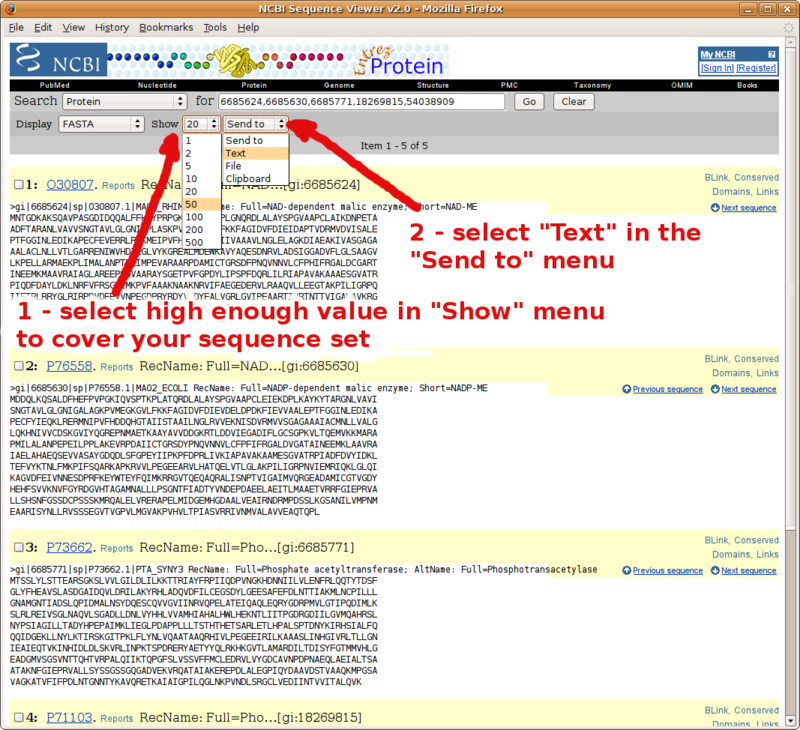
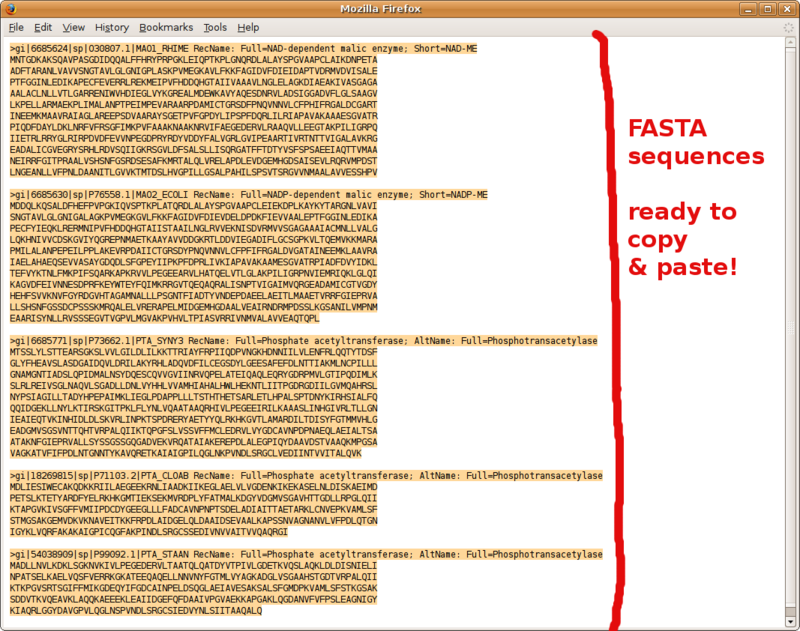
Utiliser BLAST pour compiler une liste au format FASTA des séquences homologues
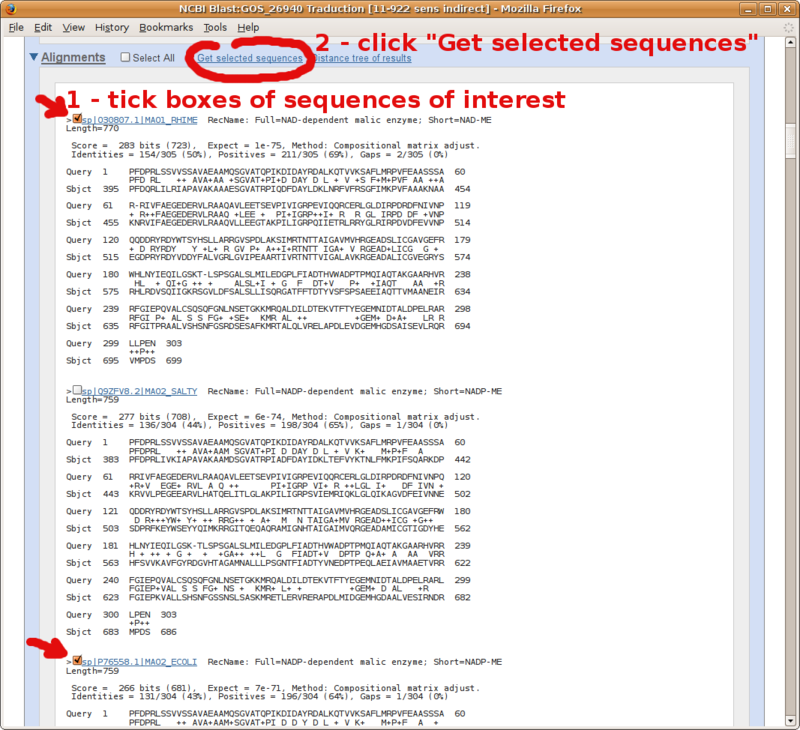
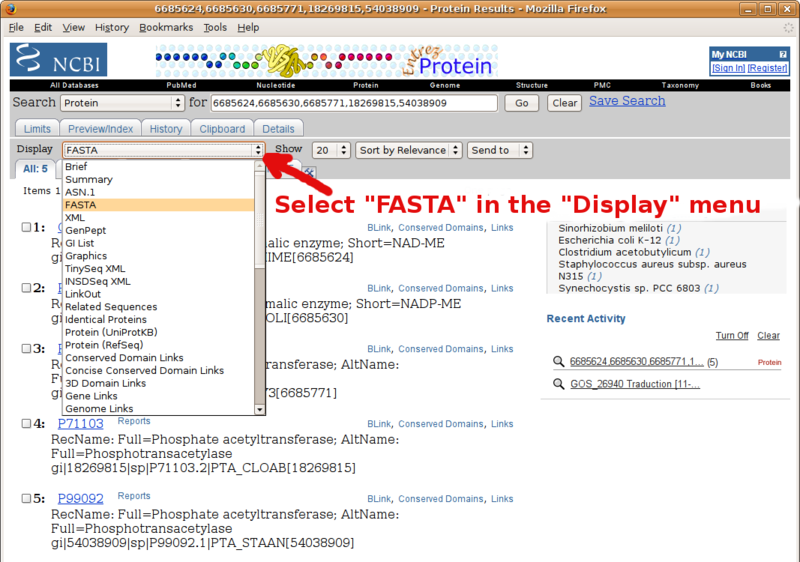
Profitez de votre page de résultats BLAST pour préparer rapidement un jeu de séquences au format FASTA (par exemple vos groupes d'étude et externe pour envoi à l'alignement multiple et la reconstruction d'arbre phylogénétique).
Rendez-vous à la section des alignements deux-à-deux de votre page de résultats BLAST (NCBI seulement), et suivez ce tutorial en images.
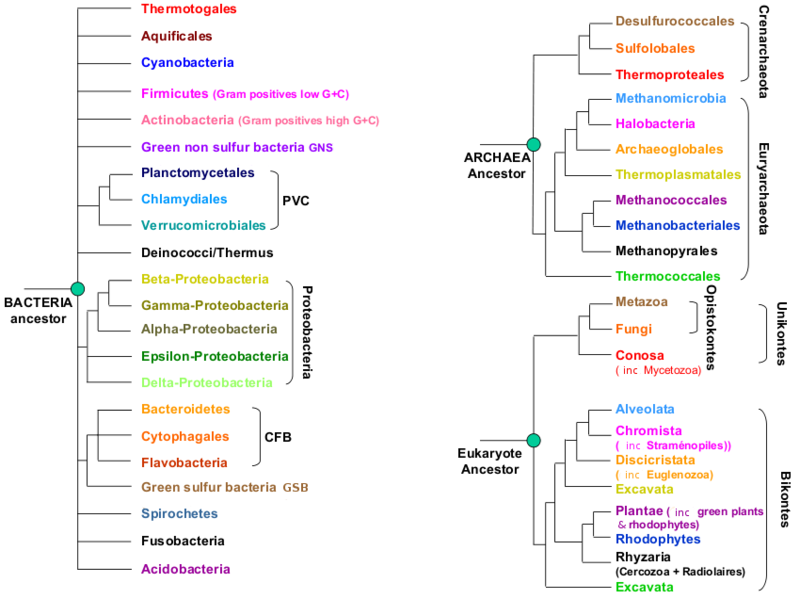
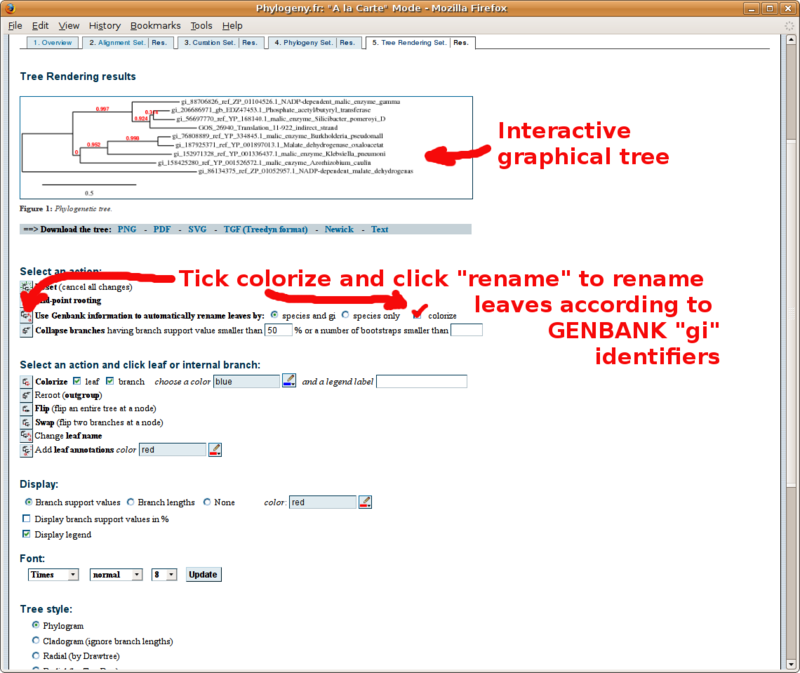
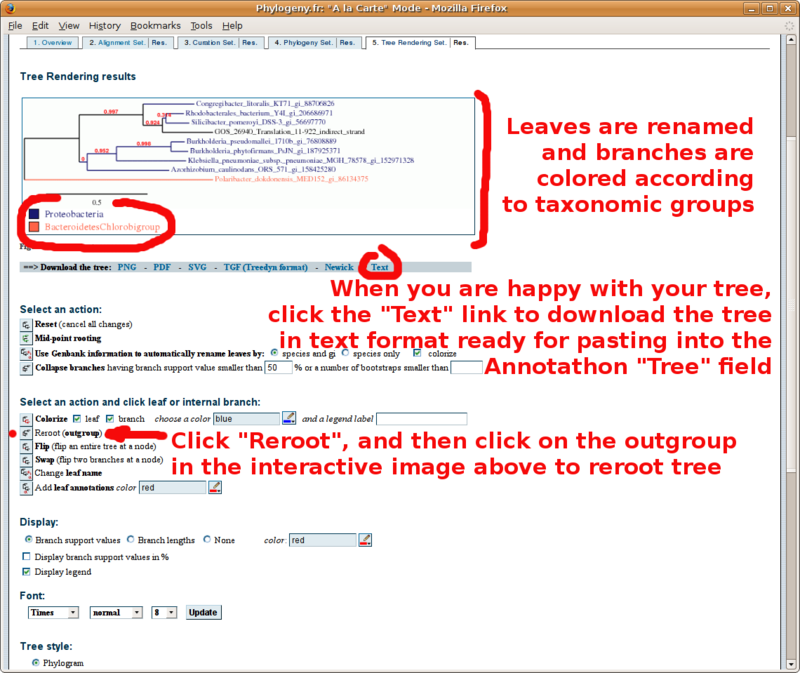
L'arbre de la vie, vu par les microbes
Il est essentiel que les annotateurs de séquences métagénomiques gardent cette vue simplifiée de l'arbre de la vie en tête en permanence. Se référer aux embranchements des branches de cet arbre est indispensable pour correctement définir ses groupes d'étude et groupes extérieurs! Vous pourriez imprimer cette image, ou même en faire votre fond d'écran...
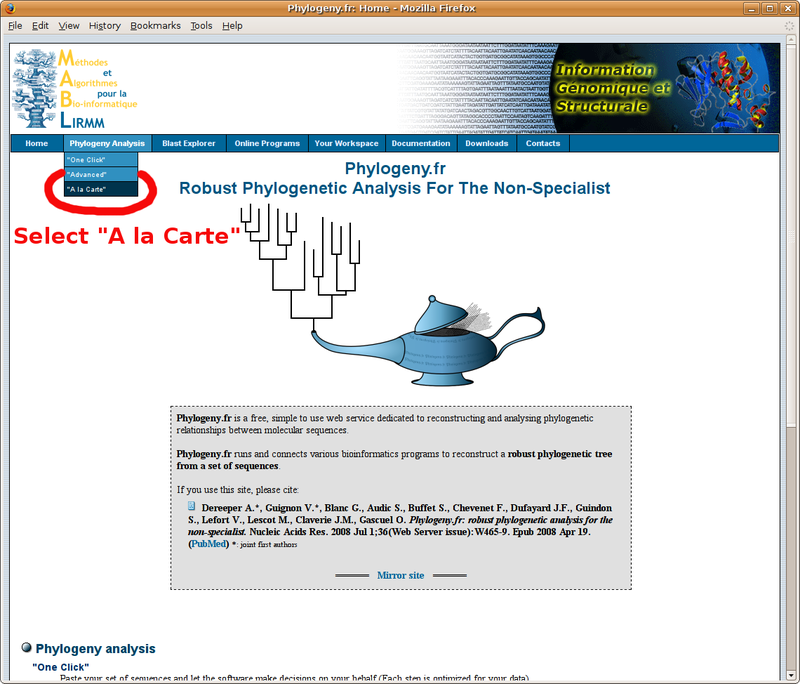
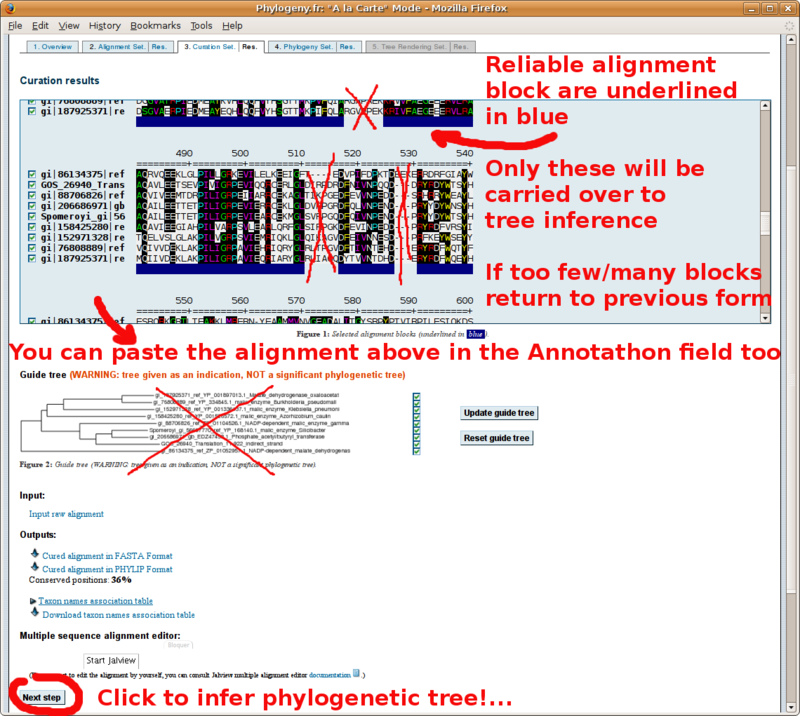
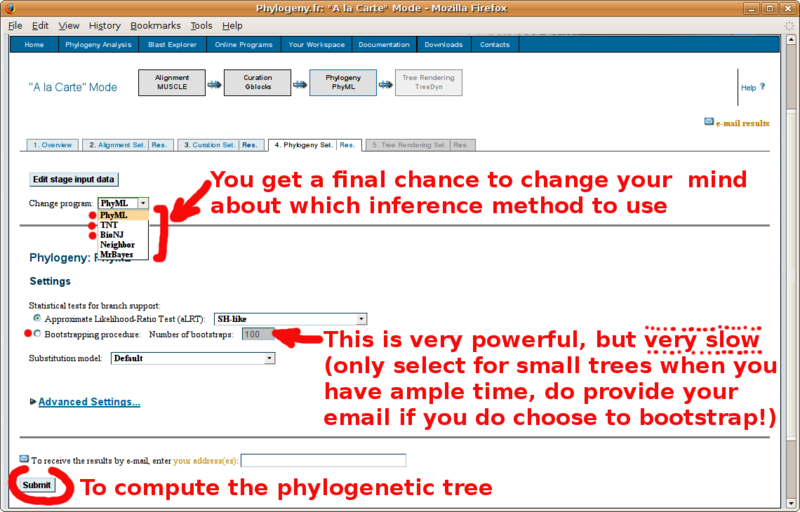
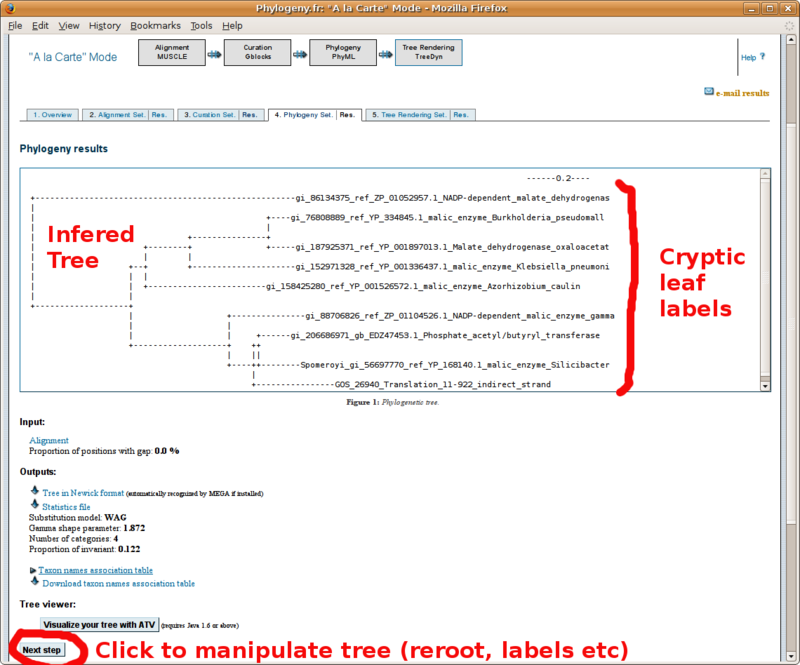
Comment reconstruire un arbre phylogénétique?
Avant d'aller plus loin, consultez ABSOLUMENT le ![]() guide de sélection des groupes d'études et extérieurs
guide de sélection des groupes d'études et extérieurs  pour la reconstruction d'arbre phylogénétiques! Ce guide donne aussi de précieux conseils sur l'interprétation des arbres obtenus.
pour la reconstruction d'arbre phylogénétiques! Ce guide donne aussi de précieux conseils sur l'interprétation des arbres obtenus.
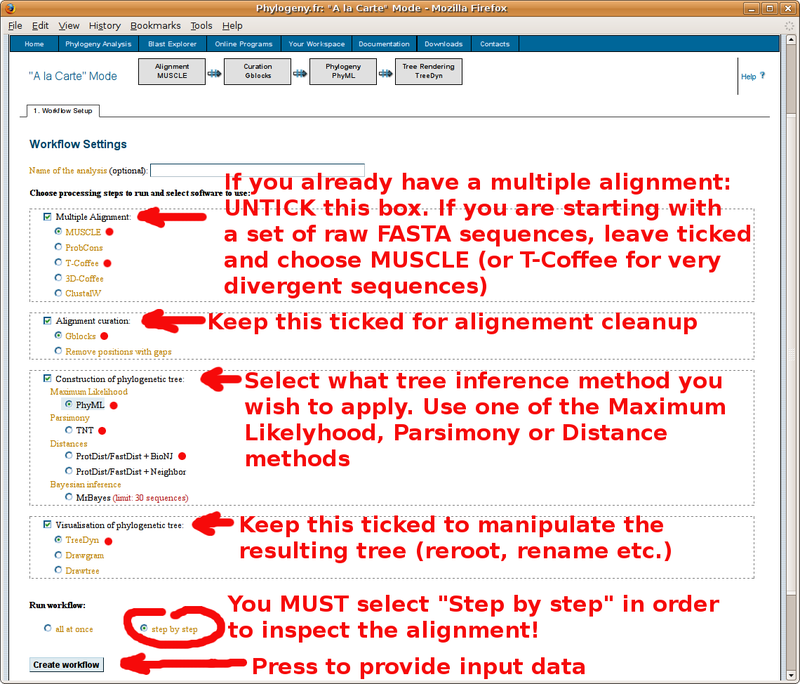
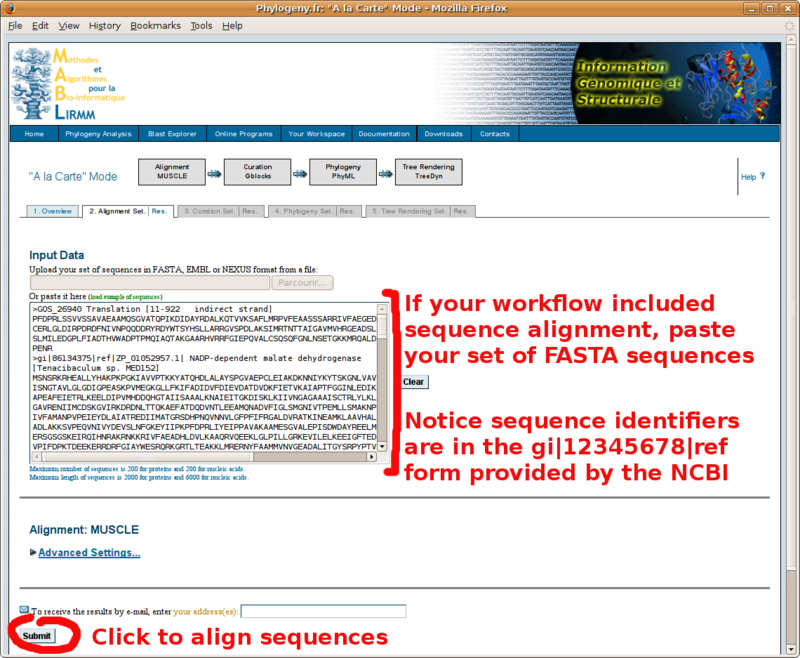
Muni de votre jeu de séquences au format FASTA, pointez votre navigateur sur http://www.phylogeny.fr/ pour construire un alignement multiple, puis un arbre phylogénétique. Vous trouverez ci-dessous un tutorial par l'image de cet outil en ligne (si vous avez déjà fait votre alignement multiple à l'EBI, ne cochez pas la case "Multiple alignment et copiez collez votre alignement à la place du jeu de séquence au format FASTA tel que présenté dans le tutorial):
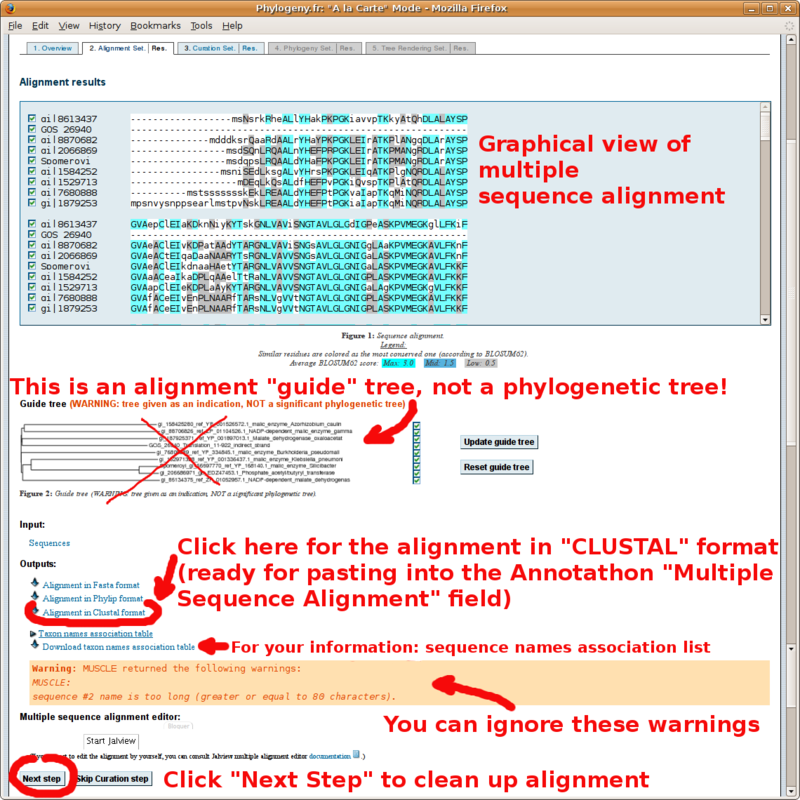
L'alignement multiple au format "MUSCLE" (identique au format CLUSTAL)
obtenu en cliquant sur le lien "Alignment in CLUSTAL format" (copiez-le
dans le champ de l'Annotathon Alignmentmultiple):
):
MUSCLE (3.7) multiple sequence alignment
gi|8613437 ------------------MSNSRKRHEALLYHAKPKPGKIAVVPTKKYATQHDLALAYSP
GOS_26940 ------------------------------------------------------------
gi|8870682 --------------MDDDKSRQAARDAALRYHAYPKPGKLEIRATKPLANGQDLARAYSP
gi|2066869 -----------------MSDSQNLRQAALNYHEFPRPGKLEIRATKPMANGRDLARAYSP
Spomeroyi -----------------MSDQPSLRQAALDYHAFPKPGKLEIRATKPMANGRDLARAYSP
gi|1584252 ----------------MSNISEDLKSGALVYHRSPKPGKLEIQATKPLGNQRDLALAYSP
gi|1529713 -------------------MDEQLKQSALDFHEFPVPGKIQVSPTKPLATQRDLALAYSP
gi|7680888 ----------MSTSSSSSSSKEKLREAALDYHEFPTPGKVAIAPTKQMINQRDLALAYSP
gi|1879253 MPSNVYSNPPSEARLMSTPVNSKLREAALDYHEFPTPGKIAIAPTKQMINQRDLALAYSP
gi|8613437 GVAEPCLEIAKDKNNIYKYTSKGNLVAVISNGTAVLGLGDIGPEASKPVMEGKGLLFKIF
GOS_26940 ------------------------------------------------------------
gi|8870682 GVAEACLEIVKDPATAADYTARGNLVAVISNGSAVLGLGNIGGLAAKPVMEGKAVLFKNF
gi|2066869 GVAEACTEIQADAANAARYTSRGNLVAVVSNGSAVLGLGNIGALASKPVMEGKAVLFKNF
Spomeroyi GVAEACLEIKDNAAHAETYTARGNLVAVVSNGTAVLGLGNIGALASKPVMEGKAVLFKKF
gi|1584252 GVAAACEAIKADPLQAAELTTRANLVAVVSNGTAVLGLGNIGPLASKPVMEGKAVLFKKF
gi|1529713 GVAAPCLEIEKDPLAAYKYTARGNLVAVVSNGTAVLGLGNIGALAGKPVMEGKGVLFKKF
gi|7680888 GVAFACEEIVENPLNAARFTARSNLVGVVTNGTAVLGLGNIGPLASKPVMEGKAVLFKKF
gi|1879253 GVAFACEEIVENPLNAARFTARSNLVGVVTNGTAVLGLGNIGPLASKPVMEGKAVLFKKF
gi|8613437 AMKLAAVHALADLAKKSVPEQVNIVYDEVSLNFGKEYIIPKPFDPRLIYEIPPAVAKAAM
GOS_26940 -----------------------------------------PFDPRLSSVVSSAVAEAAM
gi|8870682 AMQLACIDGIAALSRATTSAEAAEAYRGEQLVFGVDYLIPKPFDPRLMGVVASAVASAAM
gi|2066869 EMQIACVDGIAELARATTSAEAAAAYKGEQLNFGADYLIPKPFDPRLVAVVSSAVAKAAM
Spomeroyi AMQIACVEGIAELARITTSAEAAAAYQGEQLTFGADYLIPKPFDPRLVGVVSSAVARAAM
gi|1584252 EMKMAAVEAIAALARETPSDVVARAYGGETRAFGADSIIPSPFDPRLILRIAPAVAKAAM
gi|1529713 EMKLAAVHAIAELAHAEQSEVVASAYGDQDLSFGPEYIIPKPFDPRLIVKIAPAVAKAAM
gi|7680888 EMEIAAVNAIAELAQQEQSDIVATAYGIQDLSFGPEYLIPKPFDPRLIVKIAPAVAQAAM
gi|1879253 EMEIAAVNAIAELARQEQSDIVATAYGIQDLSFGPEYLIPKPFDPRLIVKVAPAVAKAAM
****** :..*** ***
gi|8613437 ESGVALEPISDWDAYREELMERSGSGSKEIRQIHNRAK---RNKKRIVFAEADHLDVLKA
GOS_26940 QSGVATQPIKDIDAYRDALKQTVVKSAFLMRPVFEAAS---SSARRIVFAEGEDERVLRA
gi|8870682 ETGVATRPVEDLVAYRERLDASVFRSSMIMRPVFAAAA---LSQRRIVFAEGEDERVLRT
gi|2066869 ESGVATRPIEDITAYKQKLNQTVFKSALLMRPVFEAAR---AAARRIVFAEGEDERVLRA
Spomeroyi ESGVARRPITDLEAYRQKLNQSVFKSALLMRPVFEAAA---KAARRLVFAEGEDERVLRA
gi|1584252 DTGVATRPIADFDAYNEKLDEFVFRSGFIMRPLFQRAK---QDKKRVIYAEGEDERVLRA
gi|1529713 DSGVATRPIADFDAYIEKLSEFVYKTNLFMKPIFSQAR---KEPKRVVLAEGEETRVLHA
gi|7680888 DGGVATRPIEDMEAYKVHLQQFVYHSGTTMKPVFQIARGAPAEKKRVVFAEGEEERVLRA
gi|1879253 DSGVAERPIEDMEAYEQHLQQFVYHSGTTMKPIFQLARGVEPEKKRIVFAEGEEERVLRA
: *** *: * ** * :. :. * .*:: **.: **.:
gi|8613437 AQRVQEEKLGLPILLGRKEVILELKEEIGFT----EDVPIFDPKTDEEKERRDRFGIAYW
GOS_26940 AQAVLEETSEVPIVIGRPEVIQQRCERLGLDIRPDRDFNIVNPQQD---DRYRDYWTSYH
gi|8870682 AQVIVEEMTDRPILIGRPEIIARRCEKAGLTIKPGEDFEVVNPEDD---SRHRRYWEAYL
gi|2066869 AQAILEETTETPILIGRPEVIERRCEKLGLDVRPGRDFQLVNPEND---PRYYDYWNSYH
Spomeroyi AQAILEETTETPILIGRPEVIEARCEKMGLSVRPGQDFQIVNPEND---PRYYDYWTSYH
gi|1584252 AQAVIEEGIAHPILVARPSVLEARLQRFGLSIRPGKDFEVINPEDD---PRYRDFVRSYI
gi|1529713 TQELVSLGLAKPILVGRPSVIEMRIQKLGLQIKAGVDFEIVNNESD---PRFKEYWSEYY
gi|7680888 VQIVVDEKLAKPILIGRPAVIEHRIQRYGLRLTPGVDFTIVNTEHD---ERYRDFWQTYF
gi|1879253 MQIIVDEKLAKPILIGRPAVIEQRIARYGLRLIAGQDYTVVNTDHD---ERYRDFWQEYH
* : . **::.* :: *: * :.: . * * : *
gi|8613437 ESRQRKGRTLTEAKKLMRERN-YFAAMMVNVGEADALITGYSRPYPTVIRPILESIQKDS
GOS_26940 SLLARRGVSPDLAKSIMRTNTTAIGAVMVHRGEADSLICGAVGEFRWHLNYIEQILGSK-
gi|8870682 QLMSRRGVTPDLAKVIMRTNTTAIAAIMVYCGDADSMVCGSFGQYLWHLNYVRQILAYD-
gi|2066869 KVMQRRGVTPDLAKAIMRTNTTAIGAIMVHRGEADSLLCGTFGEYRWHLNYVQQVLGGG-
Spomeroyi QLMERRGVTPDIAKAIMRTNTTAIGAIMVHRGEADSLICGTFGEYRWHLNYVEQVLGSK-
gi|1584252 EIAGRRGVTPDAARTLVRTSSTVISALAVKKGEADAMLCGIEGRFSRHLRHVRDIIGLAP
gi|1529713 QLMKRRGITQEQAQRAVISNTTVIGAIMVHRGEADAMICGTIGEYHDHYRVVQPLFGYRD
gi|7680888 KMMARKGISEQLARVEMRRRTTLIGSMLVKKGEADGMICGTISTTHRHLHFIDQVIGKRA
gi|1879253 KMMSRKGISAQMAKLEMRRRTTLIGAMLVEKGEADGMICGTVSTTHRHLHFIDQVIGKKE
. *.* : *. : . :.:: * *:**.:: * . : :
gi|8613437 GISKVAACNLMLTKQGPMFLADTTINLNPTAKDLVKISQMTSNLVKMFGMKPNVAMLSFS
GOS_26940 TLSPSGALSLMILEDGPLFIADTHVWADPTPMQIAQTAKGAARHVRRFGIEPQVALCSQS
gi|8870682 GAHPRGALSLMITEDEPLFIADTHVHPEPTPEQIADTVMAAANHVRRFGMKPNIALCSHS
gi|2066869 TYSPHGALSMMILEDGPLFIADTHVHVEPTPEQIAETVIGAARHVRRFGLAPKIALCSQS
Spomeroyi DLRPHGALSLMILEDGPLFIADTHVRSRPSPEELAEITLGAARHVRRFGIEPQIALCSQS
gi|1584252 GVRELAALSLLITPKGNLFLCDTQVQTEPNAADLAEMTILAAAHVRRFGIEPKVALLSHS
gi|1529713 GVSTAGAMNALLLPSGNTFIADTYVNHDPSPEELAEITLMAAESVRRFGIEPRVALLSHS
gi|7680888 GCSVYGAMNALVLPGRQIFLVDTHVNVDPTPAQLAEITIMAAEEVRRFGIEPKVALLSHS
gi|1879253 GAKVYAAMNALVLPNRQIFLVDTHVNVDPTPEQLAEITIMAAEEVRRFGIEPKIALLSHS
.* . :: *: ** :. *.. ::.. :: *. **: *.:*: * *
gi|8613437 NFGSTKNESSQKIREAVSYIHRNFPNAVVDGEIQADFALNPEMLAKEFPFSKLNGKKVNV
GOS_26940 QFGNLNSETGKKMRQALDILDTEKVTFTYEGEMNIDTALDPELRARLLPENR--------
gi|8870682 QFGNLDIDSGRRVRQAMALLEAREPDFAYEGEMHIDSALDPDLRARIFPNSRLQG-PANV
gi|2066869 QFGNISCDTGSRLRAAIEILDDKRRDFVYEGEMNIDTALDPELRERIFPNSRLEG-AANV
Spomeroyi QFGNQAEGSGQRLRQAIEILDSRPRDFVYEGEMNLDSALDPELRQRIFPNSRLYG-AANV
gi|1584252 NFGSNDTVCARRVRAALDILKDRAPELEVDGEMQAELALLPDARERILPHSRLQG-VANV
gi|1529713 NFGSADCPSASKMRKTLELVKARAPELMIDGEMHGDAALVESIRNDRMPDSPLKG-AANI
gi|7680888 NFGTSNAPSAQKMRDTLAILQERAPDLHVDGEMHGDVALDAALRKEILPESTLEG-EANL
gi|1879253 NFGTSNAPTAQKMRDTLAILRERAPDLQVDGEMHGDIALDANLRREVMPDSTLEG-DANL
:**. . .:* :: : :**:: : ** :* .
gi|8613437 LIFPNLESANITYKLLKEMQG-AESIGPVILGLSKAVHIVQLGASVDEMVNMAALACVDA
GOS_26940 ------------------------------------------------------------
gi|8870682 LVFAYGDAASGVRNILKMRGG-ALEVGPILMGMGNRAHIVTPSITARGLLNISALAGTDV
gi|2066869 LIFAHADAASGVRNILKMRAG-GLEVGPILMGMGNRAHIVSPSITARGLLNMAAIAGTPV
Spomeroyi LIFAHADAASGVRNVLKMKAN-GIEVGPILMGMGNRAHIVTPSITARGLLNMAAIAGTPV
gi|1584252 LVMPDLDAADIAYNMIKVLGD-ALPVGPILMGTAKPAHILGPTVTARGIVNMTAVAVVEA
gi|1529713 LVMPNMEAARISYNLLRVSSSEGVTVGPVLMGVAKPVHILTPIASVRRIVNMVALAVVEA
gi|7680888 LVLPNIDAANIAYNLLKTAAGNNIAIGPILLGAAQPVHVLTESATVRRIVNMTALLVADV
gi|1879253 LVLPNIDAANISYNLLKTAAGNNIAIGPMLLGAAKPVHVLTASATVRRIVNMTALLVADV
gi|8613437 QQREKK
GOS_26940 ------
gi|8870682 THYS--
gi|2066869 AHYG--
Spomeroyi AHYG--
gi|1584252 QSEA--
gi|1529713 QTEPL-
gi|7680888 NAVR--
gi|1879253 IAAR--
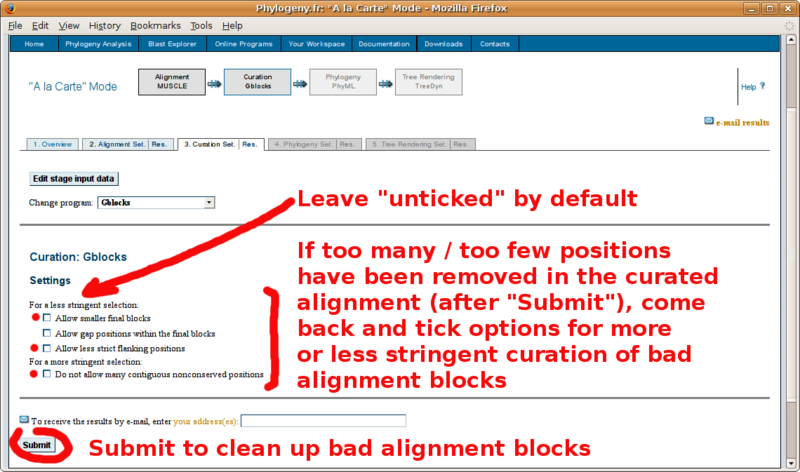
L'alignement multiple GBLOCKS après "nettoyage" (copiez-le dans le champ de l'Annotathon Alignement multiple):
Gblocks 0.91b Results
Processed file: input.fasta
Number of sequences: 9
Alignment assumed to be: Protein
New number of positions: 288 (selected positions are underlined in blue)
10 20 30 40 50 60
=========+=========+=========+=========+=========+=========+
gi|86134375|ref ------------------MSNSRKRHEALLYHAKPKPGKIAVVPTKKYATQHDLALAYSP
GOS_26940_Trans ------------------------------------------------------------
gi|88706826|ref --------------MDDDKSRQAARDAALRYHAYPKPGKLEIRATKPLANGQDLARAYSP
gi|206686971|gb -----------------MSDSQNLRQAALNYHEFPRPGKLEIRATKPMANGRDLARAYSP
Spomeroyi_gi|56 -----------------MSDQPSLRQAALDYHAFPKPGKLEIRATKPMANGRDLARAYSP
gi|158425280|re ----------------MSNISEDLKSGALVYHRSPKPGKLEIQATKPLGNQRDLALAYSP
gi|152971328|re -------------------MDEQLKQSALDFHEFPVPGKIQVSPTKPLATQRDLALAYSP
gi|76808889|ref ----------MSTSSSSSSSKEKLREAALDYHEFPTPGKVAIAPTKQMINQRDLALAYSP
gi|187925371|re MPSNVYSNPPSEARLMSTPVNSKLREAALDYHEFPTPGKIAIAPTKQMINQRDLALAYSP
70 80 90 100 110 120
=========+=========+=========+=========+=========+=========+
gi|86134375|ref GVAEPCLEIAKDKNNIYKYTSKGNLVAVISNGTAVLGLGDIGPEASKPVMEGKGLLFKIF
GOS_26940_Trans ------------------------------------------------------------
gi|88706826|ref GVAEACLEIVKDPATAADYTARGNLVAVISNGSAVLGLGNIGGLAAKPVMEGKAVLFKNF
gi|206686971|gb GVAEACTEIQADAANAARYTSRGNLVAVVSNGSAVLGLGNIGALASKPVMEGKAVLFKNF
Spomeroyi_gi|56 GVAEACLEIKDNAAHAETYTARGNLVAVVSNGTAVLGLGNIGALASKPVMEGKAVLFKKF
gi|158425280|re GVAAACEAIKADPLQAAELTTRANLVAVVSNGTAVLGLGNIGPLASKPVMEGKAVLFKKF
gi|152971328|re GVAAPCLEIEKDPLAAYKYTARGNLVAVVSNGTAVLGLGNIGALAGKPVMEGKGVLFKKF
gi|76808889|ref GVAFACEEIVENPLNAARFTARSNLVGVVTNGTAVLGLGNIGPLASKPVMEGKAVLFKKF
gi|187925371|re GVAFACEEIVENPLNAARFTARSNLVGVVTNGTAVLGLGNIGPLASKPVMEGKAVLFKKF
370 380 390 400 410 420
=========+=========+=========+=========+=========+=========+
gi|86134375|ref AMKLAAVHALADLAKKSVPEQVNIVYDEVSLNFGKEYIIPKPFDPRLIYEIPPAVAKAAM
GOS_26940_Trans -----------------------------------------PFDPRLSSVVSSAVAEAAM
gi|88706826|ref AMQLACIDGIAALSRATTSAEAAEAYRGEQLVFGVDYLIPKPFDPRLMGVVASAVASAAM
gi|206686971|gb EMQIACVDGIAELARATTSAEAAAAYKGEQLNFGADYLIPKPFDPRLVAVVSSAVAKAAM
Spomeroyi_gi|56 AMQIACVEGIAELARITTSAEAAAAYQGEQLTFGADYLIPKPFDPRLVGVVSSAVARAAM
gi|158425280|re EMKMAAVEAIAALARETPSDVVARAYGGETRAFGADSIIPSPFDPRLILRIAPAVAKAAM
gi|152971328|re EMKLAAVHAIAELAHAEQSEVVASAYGDQDLSFGPEYIIPKPFDPRLIVKIAPAVAKAAM
gi|76808889|ref EMEIAAVNAIAELAQQEQSDIVATAYGIQDLSFGPEYLIPKPFDPRLIVKIAPAVAQAAM
gi|187925371|re EMEIAAVNAIAELARQEQSDIVATAYGIQDLSFGPEYLIPKPFDPRLIVKVAPAVAKAAM
###################
430 440 450 460 470 480
=========+=========+=========+=========+=========+=========+
gi|86134375|ref ESGVALEPISDWDAYREELMERSGSGSKEIRQIHNRAK---RNKKRIVFAEADHLDVLKA
GOS_26940_Trans QSGVATQPIKDIDAYRDALKQTVVKSAFLMRPVFEAAS---SSARRIVFAEGEDERVLRA
gi|88706826|ref ETGVATRPVEDLVAYRERLDASVFRSSMIMRPVFAAAA---LSQRRIVFAEGEDERVLRT
gi|206686971|gb ESGVATRPIEDITAYKQKLNQTVFKSALLMRPVFEAAR---AAARRIVFAEGEDERVLRA
Spomeroyi_gi|56 ESGVARRPITDLEAYRQKLNQSVFKSALLMRPVFEAAA---KAARRLVFAEGEDERVLRA
gi|158425280|re DTGVATRPIADFDAYNEKLDEFVFRSGFIMRPLFQRAK---QDKKRVIYAEGEDERVLRA
gi|152971328|re DSGVATRPIADFDAYIEKLSEFVYKTNLFMKPIFSQAR---KEPKRVVLAEGEETRVLHA
gi|76808889|ref DGGVATRPIEDMEAYKVHLQQFVYHSGTTMKPVFQIARGAPAEKKRVVFAEGEEERVLRA
gi|187925371|re DSGVAERPIEDMEAYEQHLQQFVYHSGTTMKPIFQLARGVEPEKKRIVFAEGEEERVLRA
##################################### ################
490 500 510 520 530 540
=========+=========+=========+=========+=========+=========+
gi|86134375|ref AQRVQEEKLGLPILLGRKEVILELKEEIGFT----EDVPIFDPKTDEEKERRDRFGIAYW
GOS_26940_Trans AQAVLEETSEVPIVIGRPEVIQQRCERLGLDIRPDRDFNIVNPQQD---DRYRDYWTSYH
gi|88706826|ref AQVIVEEMTDRPILIGRPEIIARRCEKAGLTIKPGEDFEVVNPEDD---SRHRRYWEAYL
gi|206686971|gb AQAILEETTETPILIGRPEVIERRCEKLGLDVRPGRDFQLVNPEND---PRYYDYWNSYH
Spomeroyi_gi|56 AQAILEETTETPILIGRPEVIEARCEKMGLSVRPGQDFQIVNPEND---PRYYDYWTSYH
gi|158425280|re AQAVIEEGIAHPILVARPSVLEARLQRFGLSIRPGKDFEVINPEDD---PRYRDFVRSYI
gi|152971328|re TQELVSLGLAKPILVGRPSVIEMRIQKLGLQIKAGVDFEIVNNESD---PRFKEYWSEYY
gi|76808889|ref VQIVVDEKLAKPILIGRPAVIEHRIQRYGLRLTPGVDFTIVNTEHD---ERYRDFWQTYF
gi|187925371|re MQIIVDEKLAKPILIGRPAVIEQRIARYGLRLIAGQDYTVVNTDHD---ERYRDFWQEYH
############################## ########## ##########
550 560 570 580 590 600
=========+=========+=========+=========+=========+=========+
gi|86134375|ref ESRQRKGRTLTEAKKLMRERN-YFAAMMVNVGEADALITGYSRPYPTVIRPILESIQKDS
GOS_26940_Trans SLLARRGVSPDLAKSIMRTNTTAIGAVMVHRGEADSLICGAVGEFRWHLNYIEQILGSK-
gi|88706826|ref QLMSRRGVTPDLAKVIMRTNTTAIAAIMVYCGDADSMVCGSFGQYLWHLNYVRQILAYD-
gi|206686971|gb KVMQRRGVTPDLAKAIMRTNTTAIGAIMVHRGEADSLLCGTFGEYRWHLNYVQQVLGGG-
Spomeroyi_gi|56 QLMERRGVTPDIAKAIMRTNTTAIGAIMVHRGEADSLICGTFGEYRWHLNYVEQVLGSK-
gi|158425280|re EIAGRRGVTPDAARTLVRTSSTVISALAVKKGEADAMLCGIEGRFSRHLRHVRDIIGLAP
gi|152971328|re QLMKRRGITQEQAQRAVISNTTVIGAIMVHRGEADAMICGTIGEYHDHYRVVQPLFGYRD
gi|76808889|ref KMMARKGISEQLARVEMRRRTTLIGSMLVKKGEADGMICGTISTTHRHLHFIDQVIGKRA
gi|187925371|re KMMSRKGISAQMAKLEMRRRTTLIGAMLVEKGEADGMICGTVSTTHRHLHFIDQVIGKKE
##################### ##################################
610 620 630 640 650 660
=========+=========+=========+=========+=========+=========+
gi|86134375|ref GISKVAACNLMLTKQGPMFLADTTINLNPTAKDLVKISQMTSNLVKMFGMKPNVAMLSFS
GOS_26940_Trans TLSPSGALSLMILEDGPLFIADTHVWADPTPMQIAQTAKGAARHVRRFGIEPQVALCSQS
gi|88706826|ref GAHPRGALSLMITEDEPLFIADTHVHPEPTPEQIADTVMAAANHVRRFGMKPNIALCSHS
gi|206686971|gb TYSPHGALSMMILEDGPLFIADTHVHVEPTPEQIAETVIGAARHVRRFGLAPKIALCSQS
Spomeroyi_gi|56 DLRPHGALSLMILEDGPLFIADTHVRSRPSPEELAEITLGAARHVRRFGIEPQIALCSQS
gi|158425280|re GVRELAALSLLITPKGNLFLCDTQVQTEPNAADLAEMTILAAAHVRRFGIEPKVALLSHS
gi|152971328|re GVSTAGAMNALLLPSGNTFIADTYVNHDPSPEELAEITLMAAESVRRFGIEPRVALLSHS
gi|76808889|ref GCSVYGAMNALVLPGRQIFLVDTHVNVDPTPAQLAEITIMAAEEVRRFGIEPKVALLSHS
gi|187925371|re GAKVYAAMNALVLPNRQIFLVDTHVNVDPTPEQLAEITIMAAEEVRRFGIEPKIALLSHS
############################################################
670 680 690 700 710 720
=========+=========+=========+=========+=========+=========+
gi|86134375|ref NFGSTKNESSQKIREAVSYIHRNFPNAVVDGEIQADFALNPEMLAKEFPFSKLNGKKVNV
GOS_26940_Trans QFGNLNSETGKKMRQALDILDTEKVTFTYEGEMNIDTALDPELRARLLPENR--------
gi|88706826|ref QFGNLDIDSGRRVRQAMALLEAREPDFAYEGEMHIDSALDPDLRARIFPNSRLQG-PANV
gi|206686971|gb QFGNISCDTGSRLRAAIEILDDKRRDFVYEGEMNIDTALDPELRERIFPNSRLEG-AANV
Spomeroyi_gi|56 QFGNQAEGSGQRLRQAIEILDSRPRDFVYEGEMNLDSALDPELRQRIFPNSRLYG-AANV
gi|158425280|re NFGSNDTVCARRVRAALDILKDRAPELEVDGEMQAELALLPDARERILPHSRLQG-VANV
gi|152971328|re NFGSADCPSASKMRKTLELVKARAPELMIDGEMHGDAALVESIRNDRMPDSPLKG-AANI
gi|76808889|ref NFGTSNAPSAQKMRDTLAILQERAPDLHVDGEMHGDVALDAALRKEILPESTLEG-EANL
gi|187925371|re NFGTSNAPTAQKMRDTLAILRERAPDLQVDGEMHGDIALDANLRREVMPDSTLEG-DANL
###################################################
730 740 750 760 770 780
=========+=========+=========+=========+=========+=========+
gi|86134375|ref LIFPNLESANITYKLLKEMQG-AESIGPVILGLSKAVHIVQLGASVDEMVNMAALACVDA
GOS_26940_Trans ------------------------------------------------------------
gi|88706826|ref LVFAYGDAASGVRNILKMRGG-ALEVGPILMGMGNRAHIVTPSITARGLLNISALAGTDV
gi|206686971|gb LIFAHADAASGVRNILKMRAG-GLEVGPILMGMGNRAHIVSPSITARGLLNMAAIAGTPV
Spomeroyi_gi|56 LIFAHADAASGVRNVLKMKAN-GIEVGPILMGMGNRAHIVTPSITARGLLNMAAIAGTPV
gi|158425280|re LVMPDLDAADIAYNMIKVLGD-ALPVGPILMGTAKPAHILGPTVTARGIVNMTAVAVVEA
gi|152971328|re LVMPNMEAARISYNLLRVSSSEGVTVGPVLMGVAKPVHILTPIASVRRIVNMVALAVVEA
gi|76808889|ref LVLPNIDAANIAYNLLKTAAGNNIAIGPILLGAAQPVHVLTESATVRRIVNMTALLVADV
gi|187925371|re LVLPNIDAANISYNLLKTAAGNNIAIGPMLLGAAKPVHVLTASATVRRIVNMTALLVADV
======
gi|86134375|ref QQREKK
GOS_26940_Trans ------
gi|88706826|ref THYS--
gi|206686971|gb AHYG--
Spomeroyi_gi|56 AHYG--
gi|158425280|re QSEA--
gi|152971328|re QTEPL-
gi|76808889|ref NAVR--
gi|187925371|re IAAR--
Parameters used
Minimum Number Of Sequences For A Conserved Position: 5
Minimum Number Of Sequences For A Flanking Position: 8
Maximum Number Of Contiguous Nonconserved Positions: 8
Minimum Length Of A Block: 10
Allowed Gap Positions: None
Use Similarity Matrices: Yes
Flank positions of the 6 selected block(s)
Flanks: [402 457] [465 510] [517 526] [531 561] [564 597] [601 711]
New number of positions in input.fasta-gb: 288 (36% of the original 786 positions)
L'arbre au format "text" est copié/collé dans le champ "Arbre" de l'Annotathon (souvenez-vous d'ajouter les groupes taxonomiques pour chaque feuille):
-------0.2-----
+------------------Congregibacter_litoralis_KT71_gi_88706826 [Add taxonomic group here!]
|
| +------Rhodobacterales_bacterium_Y4I_gi_206686971 [Add taxonomic group here!]
+---------------------+ |
| | ++
| +------++--------Silicibacter_pomeroyi_DSS-3_gi_56697770 [Add taxonomic group here!]
| |
| +-----------------GOS_26940_Translation_11-922_indirect_strand
|
+------------------+ +-----Burkholderia_pseudomallei_1710b_gi_76808889 [Add taxonomic group here!]
| | |
| | +------------------+
| | +---------+ +------Burkholderia_phytofirmans_PsJN_gi_187925371 [Add taxonomic group here!]
| | | |
| | | +------------------------Klebsiella_pneumoniae_subsp._pneumoniae_gi_152971328 [Add taxonomic group here!]
| +--+
| |
| +----------------------------Azorhizobium_caulinodans_ORS_571_gi_158425280 [Add taxonomic group here!]
|
+-----------------------------------------------------------------Polaribacter_dokdonensis_MED152_gi_86134375 [Add taxonomic group here!]
A quoi sert le bloc Note?
Le Bloc Note est un champ d'annotation libre; utilisez-le pour archiver vos résultats intermédiaires, vos listes de séquences au format FASTA (avec les noms de séquence retravaillés),vos variantes d'arbres etc.